空间代谢组是基于质谱成像技术发展而来的新兴技术,通过质谱成像技术可对不同组织器官中的代谢物进行定性、定量、定位三个维度的分析,从而突破传统代谢组研究损失空间信息的瓶颈。空间代谢组学作为一种新型的分子影像技术,能够直接从生物组织中获得大量已知或未知的内源性代谢物和外源性药物等分子的结构、含量和空间分布信息[1]。
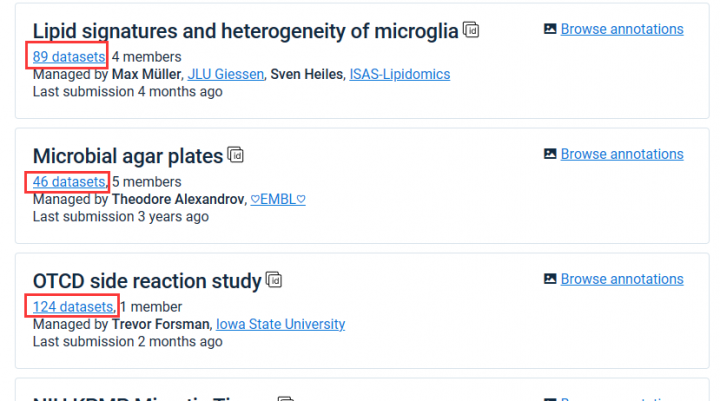
在发表SCI文章前,常规代谢组学原始数据一般上传至MetaboLights数据库(https://www.ebi.ac.uk/metabolights),但空间代谢组学的原始数据不仅有检出代谢物的原始数据,还有质谱成像的原始数据,那么空间代谢组学原始数据该如何上传呢? 基于现有文献,我们总结了获取已发表的空间代谢组学原始数据的方法,主要有以下途径:(1)通过Email直接联系文章作者获取[2,3]、(2)通过Figshare(https://figshare.com/)数据库获取(文章中说明空代数据上传至Figshare数据库)[3] 及(3)通过Metaspace数据库获取[4]等,本文重点介绍在Metaspace数据库中上传空间代谢组学原始数据的流程。
一、Metaspace数据库简介
Metascape数据库(https://metaspace2020.eu/)是一个功能强大的功能注释分析工具,能帮助用户将当前流行的生物信息学分析方法应用到批量基因、蛋白质及代谢物的分析中,以实现对基因、蛋白或代谢物功能的认知。每月更新一次,且覆盖面广泛。从数据库种类来说,Metascape整合了GO、KEGG、STRING、UniProt和DrugBank等多个权威的数据库资源,使其不仅能完成通路富集和生物过程注释,还能做基因相关的蛋白质或代谢物网络分析和涉及到的药物分析,致力于为科研工作者提供更加全面而详细的信息。同时,Metaspace数据库也是一个质谱成像数据进行代谢物注释的平台,可以用来上传空间代谢组数据。

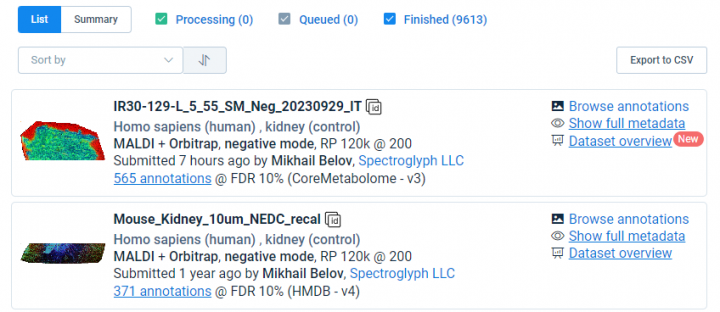
图1:Metaspace数据库首页及空代数据上传成功示例
二、Metaspace数据库可灵活暂存原始数据
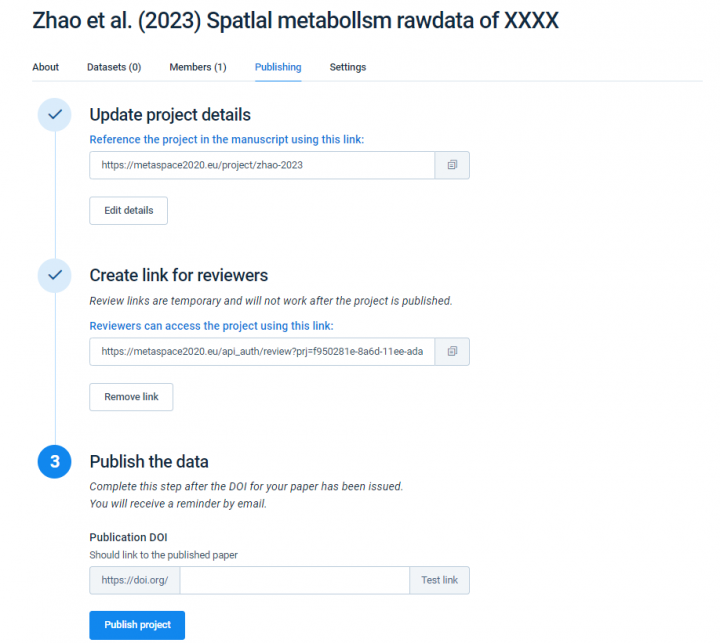
在Metaspace数据库中,可以创建一个简短的项目链接在稿件中使用,并在项目描述中添加摘要来描述项目概览。审稿人可使用审稿链接在不公开数据的情况下访问项目,有助于数据保密,并可随时撤销,论文发表后,可添加DOI并公布结果。

图2:创建临时项目链接界面
三、数据上传步骤
1.注册账号与登录
访问Metaspace网站(https://metaspace2020.eu/ ),点击右上角的Create account(新注册),或者Sign in(已有账号)。

新注册,创建账号
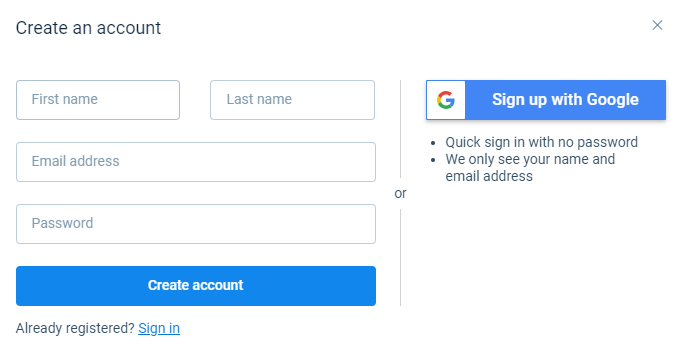
2.邮箱激活账号

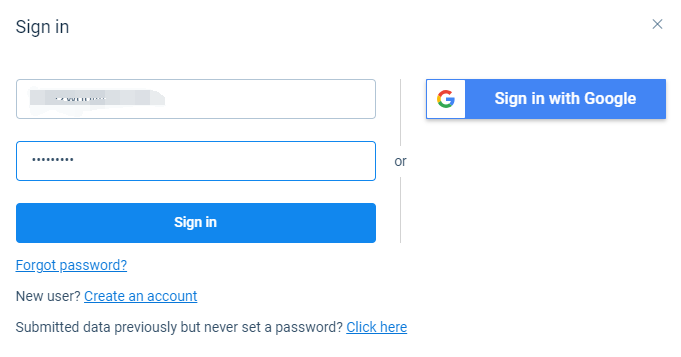
3.访问Metaspace网站,点击右上角的Sign in(已有账号)
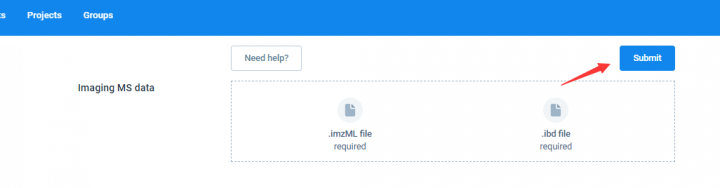
登录进去之后,点击左上角Upload或通过链接进入原始数据上传界面。空间代谢组原始数据格式为成对存在的.imzML文件和.ibd文件。单样本(单独玻片)的空间代谢组原始数据在正离子模式(Positive)或者负离子模式(Negative)应上传一次。
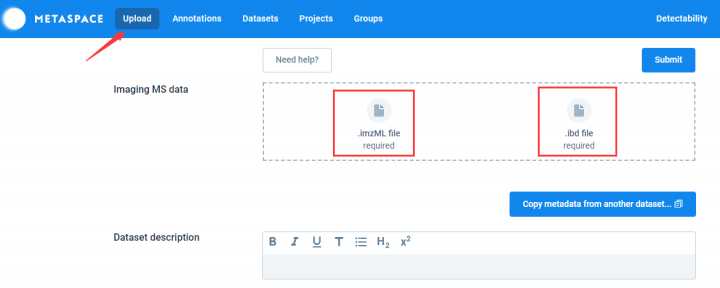
4.填写添加数据集描述和样本信息
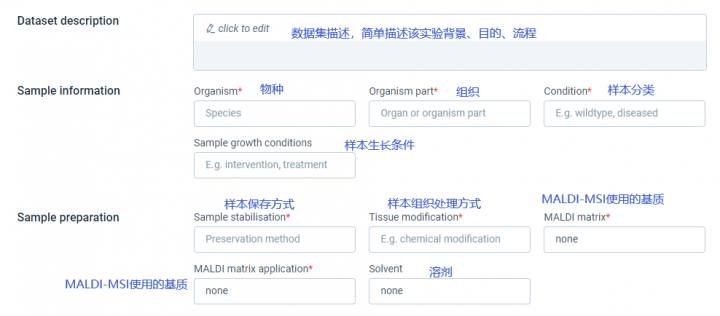
(1)Dataset description:数据集描述,简单描述该实验背景、目的、流程
(2)Organism:物种(例:Homo sapiens (human);Mus musculus (mouse);...)
(3)Organism part:组织(例:Kidney;Brain;Lung;...)
(4)Condition:样本分类(例:Wildtype;Diseased;Cancer, HCC;...)
(5)Sample growth conditions:样本生长环境(例:Surgical specimen;Caged;Cultured 2D;...)
(6)Sample stabilisation:样本保存方式(例:Fresh frozen;...)
(7)Tissue modification:样本组织处理(例:Samples were hold by the frozen (-80°C) section Leica embedding mould. && Frozen && PFA ...)
(8)MALDI matrix:这里填写MALDI-MSI使用的基质。例如DHB。
(9)MALDI matrix application:这里填写MALDI-MSI基质的使用方式,例如HTX,TM sprayer,HTX M5 sprayer
(10)Solvent:溶剂,默认none
5.根据项目情况填写分析方法
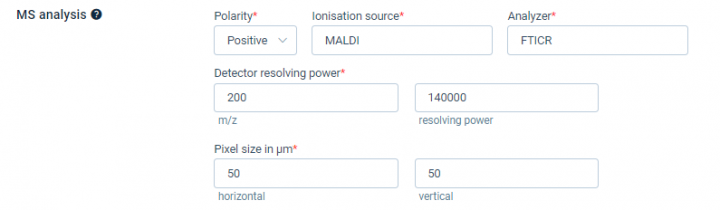
(1)Polarity:离子模式(例: negative或positive)
(2)Ionisation source:离子源(例:MALDI)
(3)Analyzer:分析平台(例:FTICR)
(4)Detector resolving power:探测器分辨能力(例:200)
(5)resolving power:解析能力(例:默认70000)
(6)Pixel size in μm:像素水平(例:horizontal 50;vertical 50)
6.填写个人信息,可以关联课题组组(Group,可不填),数据公开(Visibility,建议数据发表前勾选Private对数据保密)
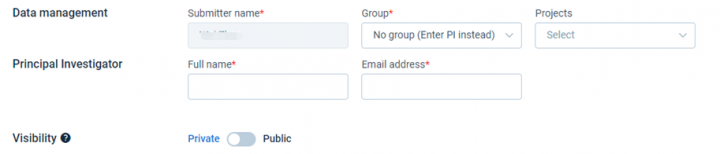
点击Projects下拉框,选择关联项目信息(Projects),在此之前需要先创建一个项目Project,如下:
在网站主页选择“Projects”,点击右侧“Create project”按钮,在弹出来的对话框中,填写项目的Title信息,如果不立即释放数据,可选择“Private”。之后点击下方“Create project”按钮创建一个新项目。
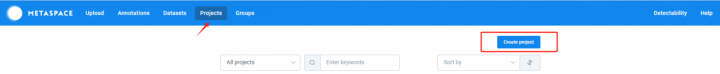
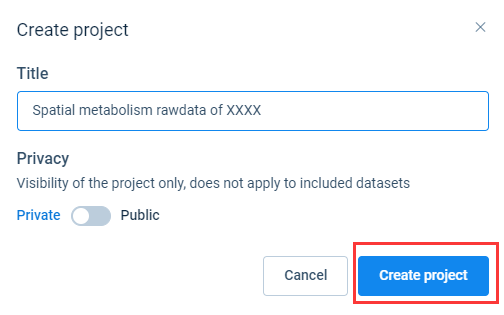
创建项目之后,填写项目描述或摘要信息

修改链接等其他信息,可用于文章中链接引用或审稿人专用临时链接,待文章发表后可添加DOI,就可以“Publish project”了
7.注释设置
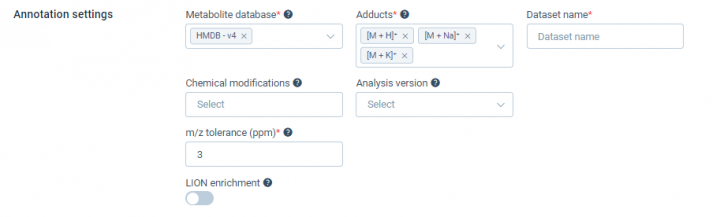
(1)Metabolite database:代谢数据库(例:HMDB,KEGG等)
(2)Adducts:离子加和形式(可以全部勾选)
(3)Dataset name:数据集命名(可为样本名)
(4)Chemical modifications:化学修饰(默认不填)
(5)Analysis version:分析版本(默认不填)
(6)m/z tolerance (ppm):ppm标准(按项目填写,建议标准为5ppm)
(7)LION enrichment:Lipid Ontology enrichment富集分析(可不勾选)
8.以上信息全部填写完毕之后就可以回到最开始点击Submit,单样本单离子模式的原始数据就上传完毕。
9.如果多个样本的空间代谢组数据上传,可在“Projects”下拉框中选择同一个项目,这些数据即可都归在该项目下。
参考文献:
[1] He J,Sun C,Li T, et al. A Sensitive and Wide Coverage Ambient Mass Spectrometry Imaging Method for Functional Metabolites Based Molecular Histology. Adv Sci (Weinh). 2018;5 (11):1800250. doi:10.1002/advs.201800250
[2] Sun C,Wang F,Zhang Y, et al. Mass spectrometry imaging-based metabolomics to visualize the spatially resolved reprogramming of carnitine metabolism in breast cancer. Theranostics. 2020;10 (16):7070-7082. doi:10.7150/thno.45543
[3] Wang G,Heijs B,Kostidis S, et al. Analyzing cell-type-specific dynamics of metabolism in kidney repair. Nat Metab. 2022;4 (9):1109-1118. doi:10.1038/s42255-022-00615-8
[4] Tu A,Said N,Muddiman DC. Spatially resolved metabolomic characterization of muscle invasive bladder cancer by mass spectrometry imaging. Metabolomics. 2021;17 (8):70. doi:10.1007/s11306-021-01819-x
免责声明:市场有风险,选择需谨慎!此文仅供参考,不作买卖依据。
文章投诉热线:182 3641 3660 投诉邮箱:7983347 16@qq.com




